近期,西南大学资源昆虫全国重点实验室的研究成果“发现调控延长多种物种寿命的新基因”入选了2024年度重庆市基础研究代表性成果。这引起研究团队认真关注了该项成果发表后的反响,整理了有关信息,以此回馈有关方面的支持和关切。
在全球老龄化趋势日益加剧的背景下,延缓衰老和延长健康寿命的科学研究显得尤为重要。2024年8月,资源昆虫全国重点实验室代方银教授团队在国际知名学术期刊Nature Communications(《自然通讯》)上发表的研究论文“FOXO-regulated OSER1 reduces oxidative stress and extends lifespan in multiple species”(FOXO调节的OSER1可减少氧化应激而延长多种物种的寿命)为健康衰老和延长寿命提供了新的科学依据,在学术界产生了重要影响,引起热烈讨论。
在出版界,单篇论文的影响力通常可以通过各种指标从不同的侧面进行衡量,包括引用次数、下载量、社交媒体讨论、Altmetric指数等。其中,Altmetric是一种综合性的学术论文影响力衡量指标,它考虑了论文在社交媒体、新闻媒体、博客、政策文件等多个非传统学术渠道中的提及和讨论情况,是对基于引用性指标的重要补充。Altmetric分数通常包括论文在Twitter、Facebook等平台上的引用情况。这种指标在于提供一种更全面的视角来评估学术论文的社会影响力,而不仅仅是学术领域的引用次数,因此,在国外的一些单位也被用作评价论文影响力的重要指标之一。《Nature》系列刊物作为国际知名的学术期刊,其在官方主页公布的每篇学术论文上均附带有表征该篇论文影响力的Altmetric数值。作为衡量科研成果影响力的重要标准之一,通常Altmetric指数达到10以上,被认为是具有高影响力的论文。
代方银教授为最后通讯作者、宋江波副教授为第一作者的这一学术论文,自在Nature子刊Nature Communications发表以后,迅速在国际学术界引起广泛关注,其Altmetric指数目前超过了280,位于涵盖超过2700万出版物(科技论文)的影响力榜单前5%,同期出版物影响力前1%。

图1 研究成果的Altmetric指数图示
与同领域研究相比,该论文在深度和广度上都显示出了自身的优势。研究团队不仅在家蚕、线虫、果蝇等多种生物模式中揭示了OSER1基因的延寿作用,还在人类受试者中发现了与长寿相关的基因变异,这一跨物种的研究在衰老和寿命调控机制研究中呈现了新的范式。在同期发表的论文中,该项工作因突出的创新性和科学价值而引人瞩目;在Nature Communications同系列期刊中,该论文也因其高质量的研究工作和重要的发现而受到特别关注。
在国外,该成果受到了包括丹麦国家电视台、美国科学促进会(AAAS)旗下科学媒体EurekAlert、科学综合媒体Science Daily、医学媒体Medical Xpress和News-Medical、抗衰老领域专业媒体Fight Aging的亮点报道。此外,还受到众多国家的官方媒体报道,如俄罗斯医学媒体medvestnik、墨西哥媒体SIPSE、葡萄牙媒体Zap.aeiou.pt、法国媒体nouvelles-du-monde;同时,波兰、韩国和日本等国媒体也进行了相关报道,凸显出该研究的重要国际影响力。
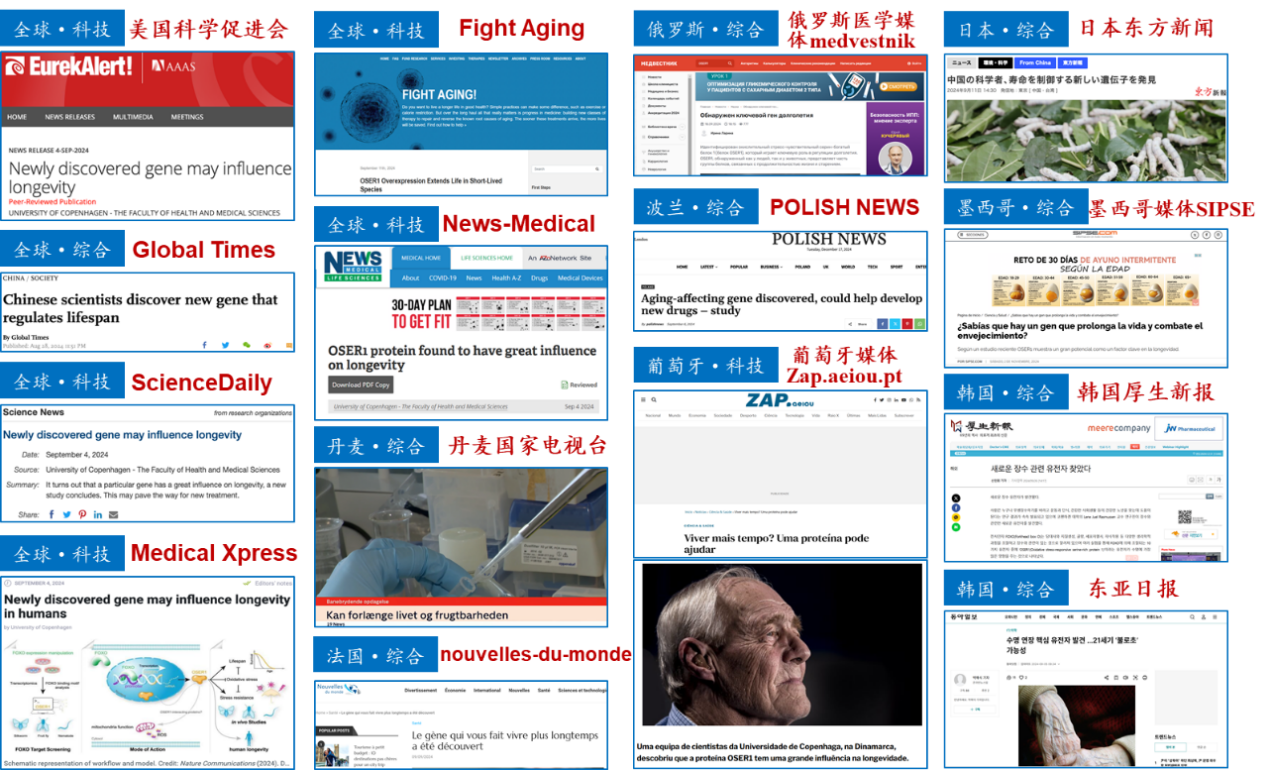
图2 报道了该研究成果的国际媒体
国内新闻媒体也广泛报道了这一研究成果。人民日报、人民日报海外版、光明日报、央视网、人民网、中国科学报、重庆日报等媒体对该研究成果进行了重点报道。全网搜索显示,关于该论文的报道,覆盖了科技、健康、教育等多个领域。
研究团队通过精细的实验设计,在基于组学分析和筛选FOXO靶基因的基础上,通过一系列的实验研究,包括基因敲低、过表达、蛋白质组学分析等方法,揭示了OSER1基因在延缓衰老和延长健康寿命过程中的关键作用和物种间的保守性。尤为重要的是,该研究在人类受试者中发现了OSER1基因与长寿相关的变异,为未来针对该基因的抗衰老干预提供了潜在的分子靶点。
国际学术同行对该项研究成果给予了高度评价。其中,美国约翰霍普金斯大学(医学领域世界排名并列前二)的首席研究员Benjamin Orsburn给予了热情洋溢的评价,他表示:该研究深入挖掘了衰老机制,是近两年来该领域尤为突出的工作。以下是其评价的原文译文:
“我一点也不轻视Nature Communications,但直到我从截取的该论文PDF看到期刊名称前,我真的以为是Nature上的论文。后者看来对衰老机制研究情有独钟(我也一样,这不仅仅是因为我最近的研究资助来源,变老实在是一件糟糕的事情,我希望自己比现在老得更慢一点)。而这一篇论文是我在过去一两年里读到的最完整的论文。在Nature系列期刊上高调发表的衰老研究中,与最近的许多研究相比,这项研究尤其突出。他们提供了所在国家和大陆法律许可范围内的所有数据,甚至这些数据可以通过提出申请来获取,就像从CPTAC联盟获取患者肿瘤基因组数据一样;并且这项研究提供了海量的数据,蛋白质组学只是其中微不足道的一部分,事实上,在方法部分描述的第34个实验才是蛋白质组学。
他们在众多模式生物中观察了靶蛋白的敲低和过表达情况。他们跟踪统计了这些生物的整个生命周期,并发现靶蛋白含量增多会延长寿命,反之则缩短寿命。这项研究采用了高精度显微观察、活性氧检测和转录组学等方法,并通过qPCR/RT-PCR对结果进行验证。——此外在人类细胞进行转染,并获得了其蛋白组数据。
接下来(注意听好了)
他们观察了一群显然非常酷的90多岁老人,他们就像在说‘没错,你瞧瞧我为什么还能在球场上碾压那些75岁的家伙’,然后他们对序列变异进行了研究。
他们真的有所发现!这是你只点击论文中的链接无法获取的数据。单核苷酸多态性(SNPs)并不是绝对的‘大获全胜’,但似乎确有端倪。如果你考虑到所有人在魔术贴被发明之前就活得精彩纷呈,经历了各种环境暴露、饮食差异和人生经历,那么从一个基本上完全未知的蛋白质(你去查查,会发现一无所获)的单核苷酸多态性中发现某些东西,可是一件了不起的大事。
我可以继续谈论这项鼓舞人心的工作以及它是一个多么完整和令人激动的故事。只要你不看补充材料和源数据,这就是一篇简短的阅读。既然我不想让你认为我作为一个爱挑刺的批评博主已经失去了水准,我不得不指出源数据有点难以跟上。所有内容都在那里,但我不得不在图表和表格之间来回切换数次以弄清楚我在看什么(或者向下滚动很远)。
再次强调,这只是我在挑毛病,因为这无疑是我最近读过的最喜欢的研究。
这个团队太棒了,我非常期待他们正在进行的后续研究。
作者:本”
附:Benjamin Orsburn评述该项研究的英文全文(https://proteomicsnews.blogspot.com/):
FOXO-regulated OSER1 extends lifespan in a bunch of species!

I mean exactly zero slight to Nature Communications, but until I took the above screenshot from the PDF, I really assumed this was one word Nature. The latter seems to really dig aging mechanism studies (as do I, and not just because of my recent funding sources, getting old is dumb and I'd prefer to do less of it than I currently am) and this is the most complete work I've read in the last year or two.
On the note of high profile aging studies in one word journals, where this one particularly shines in comparison to a whole lot recently is here
They provided all of the data that they are legally allowed to under the laws of their country and their continent. And even that can be obtained by filing a request to access it, just like getting patient tumor genomics data from the CPTAC consortium.
AND this study an absurd amount of data. Proteomics is the tiniest part of it.
In fact - the 34th experiment described in the method section - is the proteomics.
They looked at knock-downs of the target protein and overexpression in a slew of model organisms. They followed these organisms over their lifespan and found that more of it increases lifespan and less does the opposite. There is fancy microscopy and ROS assays and transcriptomics- and confirmation of those results with qPCR/RT-PCR and then they transfected human cells and that's where the proteomics comes in.
THEN (Get this)
They looked at a cohort of obviously super cool people in their 90s who are like "hell yeah, you can see why I'm still crushing the 75 year olds on the pickleball court" and they looked at sequence variants.
And they f'ing find some! That's the data you can't have by just clicking on the link in the paper. The SNPs aren't an absolute homerun, but it looks like something is there. And if you consider all of the environmental exposures and differences in diet and experiences that people have were hanging out on this planet being cool before velcro was invented seeing something from single nucleotide polymorphisms of a protein that is basically completely unknown (look it up, you'll get a bunch of nothing) before this paper is a huge deal.
I could keep going on about what an inspirational work and what a complete and exciting story it is. It's a short read as long as you stay out of the supplemental and source data. And since I don't want you to think I've slipped off of my game as a cantankerous critical blogger, the source data is a little tough to follow. It's all there, but I had to go back and forth between figures and sheets to a couple times to sort out what I was looking at (or scroll way way down).
Again, that's just me being a jerk as this is by far my favorite thing I've read recently.
Bravo to this team and I'm super pumped for the follow-up studies they're clear are in progress.
Posted by Ben
(供稿:资源昆虫全重室 宋江波; 审核:代方银)