2023年11月6日,生物学领域国际著名期刊Nucleic Acids Research(《核酸研究》)在线发表了西南大学资源昆虫高效养殖与利用全国重点实验室代方银教授团队的最新科研成果——家蚕泛基因组和多组学数据分析平台(SilkMeta)。这是继2022年研究团队发表家蚕泛基因组论文(Nat. Commun., 2022)之后,在家蚕种质资源基因组和生物信息分析领域取得的又一突出进展,标志着团队创建“数字家蚕”的工作迈入新阶段,对于推动家蚕泛基因组成果在功能基因组、基因组选择和分子设计育种等方面的研究应用具有重要意义。

由于家蚕大规模种质资源泛基因组的完成,家蚕组学数据真正海量式增加,推动家蚕重要性状遗传基础解析和分子育种研究步入快速发展阶段。然而,多组学大数据的整合分析和使用具有成本高、技术难度大的特点,这也是横亘在大数据和科研工作者之间的一座高墙。为破解这一阻碍,团队着力开辟解析表型到基因型关系的快速通道,构建了家蚕泛基因组和多组学数据库——SilkMeta(http://silkmeta.org.cn/)。
SilkMeta整合了家蚕基因组、转录组、变异组、表型组等多组学数据和功能基因研究数据,并提供了开展比较基因组和功能基因组等方面研究的分析工具。目前数据库涵盖了丰富的数据内容,包括:294种家蚕质量性状基因型特征、546个基因组、1168个转录组、4300多万个单核苷酸变异(SNPs)、930多万个小片段插入/缺失(InDels)、340多万个大片段结构变异(SV)、19411个种内基因簇、7216个种间基因簇以及家蚕的全长转录本、转座子、转录因子、small RNA(circRNA、miRNA、piRNA)、染色质免疫共沉淀测序(ChIP-seq)数据、甲基化测序数据、Hi-C测序数据等,是迄今涵盖数据类型最全、数据量最大的家蚕组学数据库。
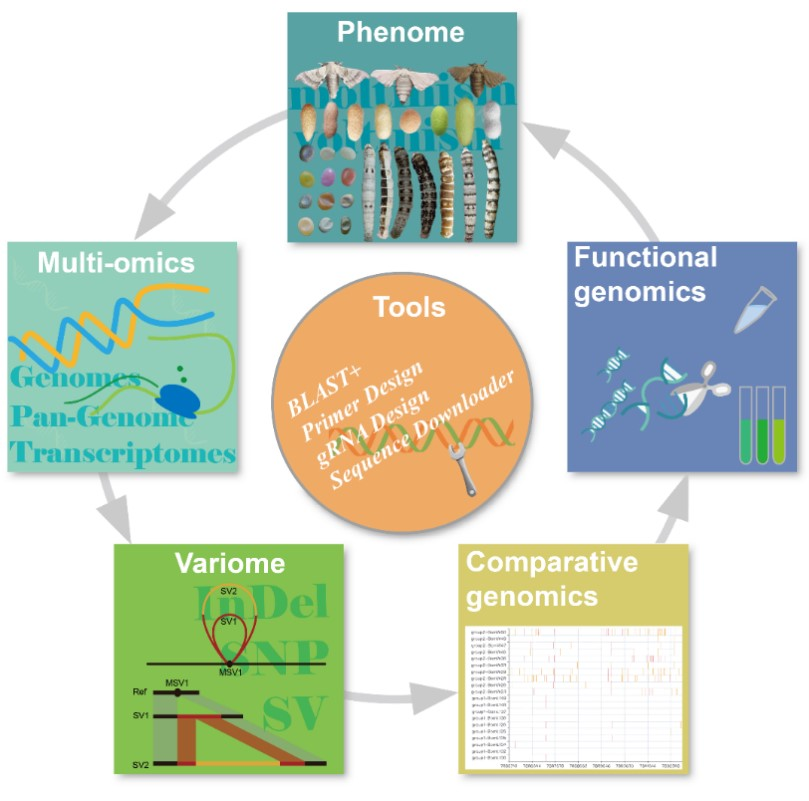
为了使SilkMeta中多组学数据的可视化和分析更便捷,研究团队开发了12个功能模块,包括:样本信息(sample information)、群体结构(population genetics)、基因搜索(gene search)、变异搜索(variation search)、变异可视化(variations viewer)、基因组浏览器(genome browser)、基因表达(expression)、选择性清扫(selective sweep)、功能基因研究(functional research)、突变图谱(mutant map)、工具模块(tools: Blast、引物设计、gRNA设计、序列提取)和数据下载(download)页面。
其中,样本信息模块提供了家蚕各品系的基本信息(样品编号、名称、性别、地理来源、表型描述等)、地理分布情况和二代、三代测序信息,基因组注释信息等;群体结构模块展示了家蚕各品系的系统发生关系和聚类情况;基因搜索工具提供了基于101个基因组的基因搜索和基因的序列、结构、结构域注释、同源基因(种内和种间)、基因分类(核心基因、次核心基因、可变基因和私有基因)、关联文献等信息;变异搜索工具提供了基于基因和基因组位置的两种搜索模式,用户可获得搜索范围内变异(SNP、InDel、SV)的等位基因频率(包括在野桑蚕、地方种、实用种各亚群中的等位基因频率)、功能注释等信息;通过变异可视化模块,用户可以比较一个基因组区域内的变异在多个样本/群体中的分布情况;基因表达模块整合了已公布的122个项目1168个转录组数据,覆盖家蚕的43个组织和48个不同发育时期,用户可在此模块中输入一个或多个基因编号,选择感兴趣的组织、发育时期或研究项目以查看基因的表达模式;基因组浏览器展示了基因结构信息、变异信息、非编码RNA、小RNA、ChIP-seq的峰值等;在选择性清扫模块中,用户可以查看不同群体间的选择性清扫信号,判断候选基因或基因组区域是否存在人工选择;在突变图谱模块,展示了家蚕遗传连锁图谱及相关研究进展;基因功能研究模块整合了现有的家蚕功能基因研究进展文献……
通过SilkMeta平台,用户可以全面快速挖掘表型关联基因/变异(特别是大片段结构变异),品系稀有/特有变异,结合家蚕遗传连锁图谱和物理图谱,高效精准锁定特定性状的关联位点/基因;可开展基因的表达模式分析、选择压力分析、调控位点分析和功能分析等;可利用变异数据延伸开展数量性状全基因组关联分析(GWAS)和育种芯片设计。为显示平台在基因/变异挖掘中的作用,研究团队复现了一个家蚕突变体(Bo)控制基因(变异)和一个蚕卵孵化率相关基因(SP1)及其关联变异的挖掘过程,详细分析步骤可在用户手册(http://silkmeta.org.cn/help/userManualGeneSearch)中查看。
作为一个综合性家蚕组学数据库和分析平台,SilkMeta将得到持续更新,为家蚕及昆虫学等领域研究者提供丰富的生物信息资源和分析工具,加速家蚕生物学基础研究和现代育种。未来,研究团队将继续整合新的组学数据和有关研究进展,不断充实和优化SilkMeta的数据资源和用户体验。
西南大学为该论文的第一署名和通讯单位。资源昆虫高效养殖与利用全国重点实验室博士后陆昆鹏为论文第一作者,代方银教授为论文最后通讯作者。该研究由国家自然科学基金重点项目、现代农业产业技术体系项目、重庆市自然科学基金创新群体项目等提供经费支持。
论文链接:家蚕泛基因组和多组学数据分析平台(SilkMeta)
(供稿:陆昆鹏 审核:代方银)