| 发布时间:2023-07-25 查看 0次 字体:[大] [中] [小] |
桑树是蔷薇目桑科桑属的重要经济林木,在世界范围内广泛分布。桑树具有丰富的遗传资源,其不仅是全球养蚕业的基础,更对人类的健康和生态环境保护具有重要作用。2013年首个基于二代测序技术的桑树基因组发表,极大地推进了桑树遗传育种和种质资源改良进程。2022年该团队又基于三代ONT技术重新测序组装新版基因组,使得基因组的完整性进一步提高。然而该基因组与预估的基因组大小依然有较大差距,依然存在未组装完成的序列以及未挂载到染色体的序列,这些序列中包括了着丝粒、端粒以及其他一些重复序列,序列的缺失限制了对基因组的利用。

2023年5月,国际著名学术期刊Horticulture Research上线西南大学桑树研究团队题为“The gap-free genome of mulberry elucidates the architecture and evolution of polycentric chromosomes”的研究论文。这是桑树首个报道的无缺口参考基因组,是继2013年桑树基因组图谱发表后,桑树基因组学领域中又一里程碑事件。

该研究基于川桑Morus notabilis,利用PacBio HiFi、ONT ultra-long以及Hi-C技术组装得到首个0 gap的桑树完整基因组Mnot-SWU。该基因组大小为410.45 Mb,contig N50达到75.38 Mb,BUSCO评估基因组完整性为98.51%。通过注释,共获得27413个基因,基因数量高于之前所有基因组版本中基因数量。在该Mnot-SWU基因组中,重复序列占比为59.20%。
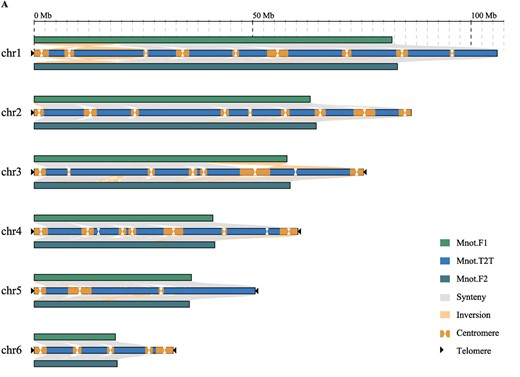
Figure 1. 桑树T2T基因组(Mnot-SWU)
作者开展比较基因组分析,发现与之前版本基因组相比,Mnot-SWU共新组装出83.81 Mb的区间,Mnot-SWU的每条染色体上均存在数个新组装的区间,且这些新组装区间与基因组中串联重复序列分布模式一致,与基因分布模式相反。作者进一步通过对串联重复序列聚类鉴定出桑树的着丝粒序列m3cp,通过分析m3cp的分布模式,并进一步结合Chip-seq以及FISH验证,最终证实桑树是一个多着丝粒染色体的物种。通过对chr5染色体结构特征的分析,提出了桑树染色体断裂融合循环的模型:在chr5染色体的28-33 Mb区间内存在一个新形成的着丝粒,该着丝粒的形成,介导了桑树染色体的断裂融合现象。
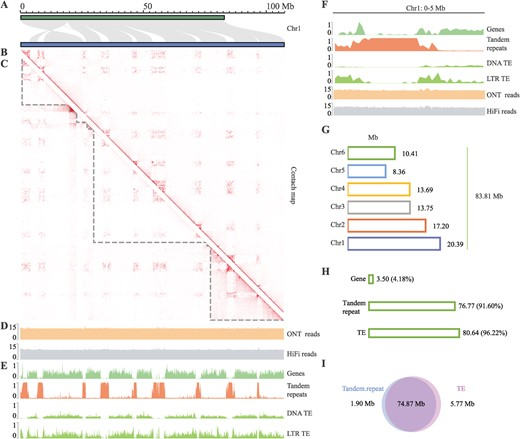
Figure 2. 桑树染色体被富含重复序列的区域分成多个区间

Figure 3. 桑树多着丝粒染色体的特征
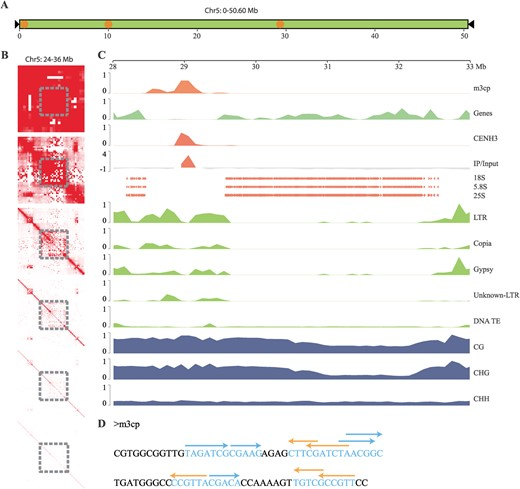
Figure 4. chr5染色体的结构分析
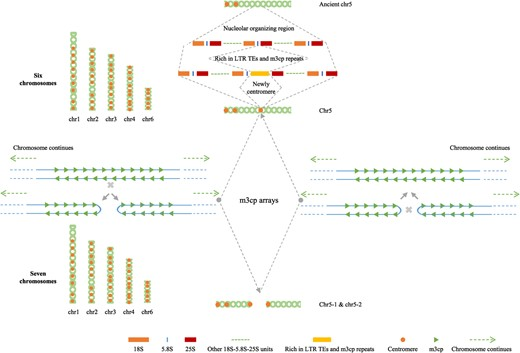
Figure 5. 桑树染色体断裂融合循环的模型
在2013年首个桑树基因组发布10年后,该研究公开了桑树T2T完整参考基因组,这是植物中首个报道的多着丝粒形态的T2T基因组,不仅为桑树染色体的进化研究提供重要参考,更对桑树的育种和遗传改良具有重要意义。
西南大学资源昆虫高效养殖与利用全国重点实验室马赑副教授为论文第一作者,研究生王虹虹、刘京纯、陈林、夏小雨、魏吴琦、杨震以及青年教师袁姜莲、罗义维均参与了该项工作,何宁佳教授为论文通讯作者。本研究工作得到国家自然科学基金和重庆市基础研究与前沿探索类项目资助。