西南大学资源昆虫高效养殖与利用全国重点实验室主任、国家蚕桑产业技术体系首席科学家代方银教授团队基于前期成果“千蚕泛基因组”研发首款家蚕全基因组SNP育种芯片—Silk_40K SNP Array,首次证明基于育种芯片的基因组选择可实现家蚕复杂育种性状的高效改良,为家蚕基因组选择育种提供了关键工具与理论支撑。2025年11月14日,研究成果在线发表于国际知名期刊Journal of Advanced Research(中科院一区Top,IF=13)。

长期以来,家蚕育种以传统的杂交选育及系统分离选择为主,对茧丝产量、品质等复杂育种性状的改良早已进入平台期。突破家蚕育种瓶颈对包括基因组选择在内的先进技术具有根本性依赖,但此前尚缺乏低成本、高通量、高重复性的基因分型工具。为突破这一障碍,研究团队依托前期构建的家蚕超级泛基因组图谱和SilkMeta数据库,精心筛选40,887个覆盖全基因组的代表性SNP为背景位点集,整合包括家蚕茧丝产量关联基因、驯化及育种改良效应基因在内的1000个功能前景位点,成功开发出家蚕首套全基因组SNP育种芯片Silk_40K Array。
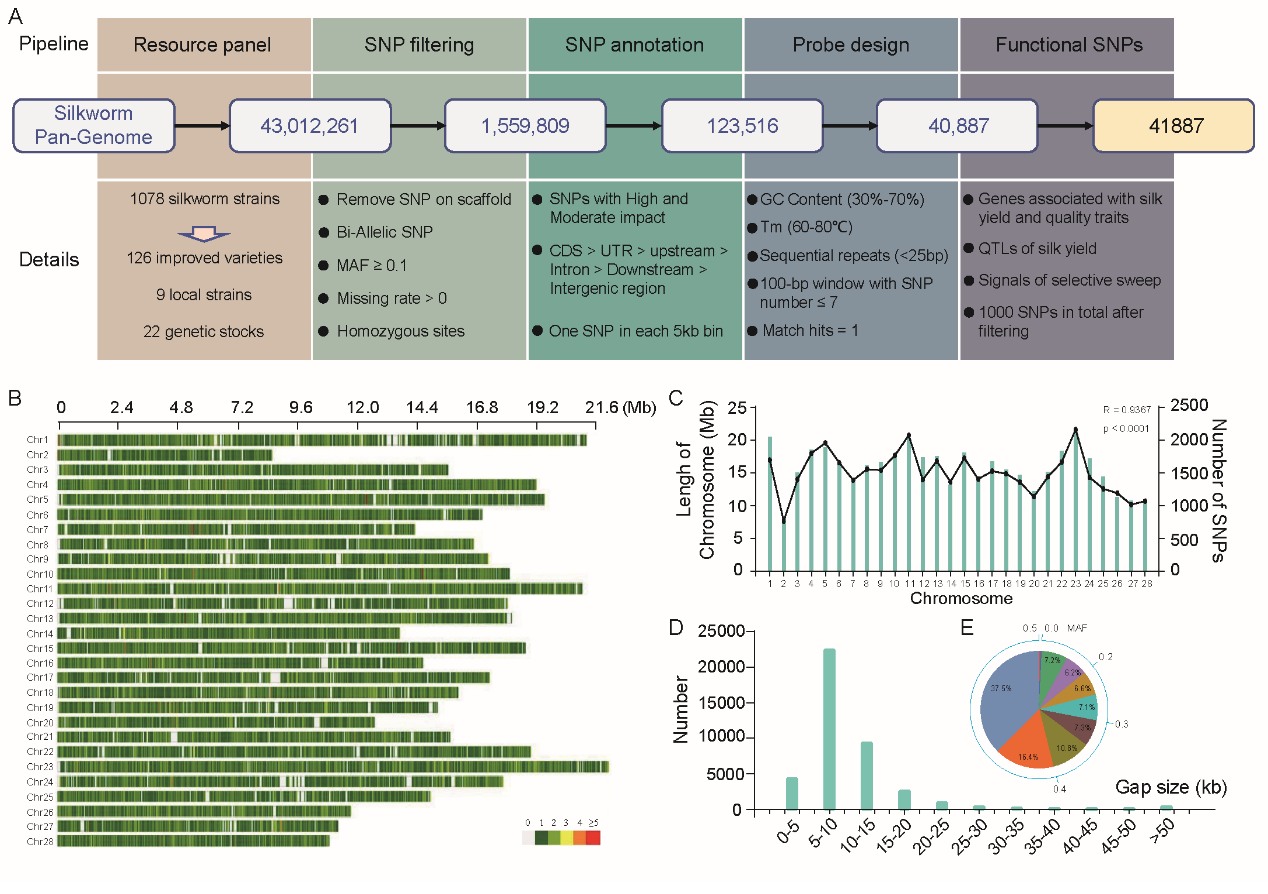
Silk_40K Array设计流程及位点分布特征
与全基因组SNP对比显示,芯片位点分布均匀、位点密度高(位点间平均间距20.9kb),且富含更高比例的高效应位点。在12个种质资源样本中的分型结果显示,芯片具有分型准确(平均检出率94.01%,最高达96.58%)、多态性水平高(平均多态率37.42%)等优势,且分型结果高度可重复,重复分型一致性均高于99%。充分证明了Silk_40K Array稳定高效的分型能力。
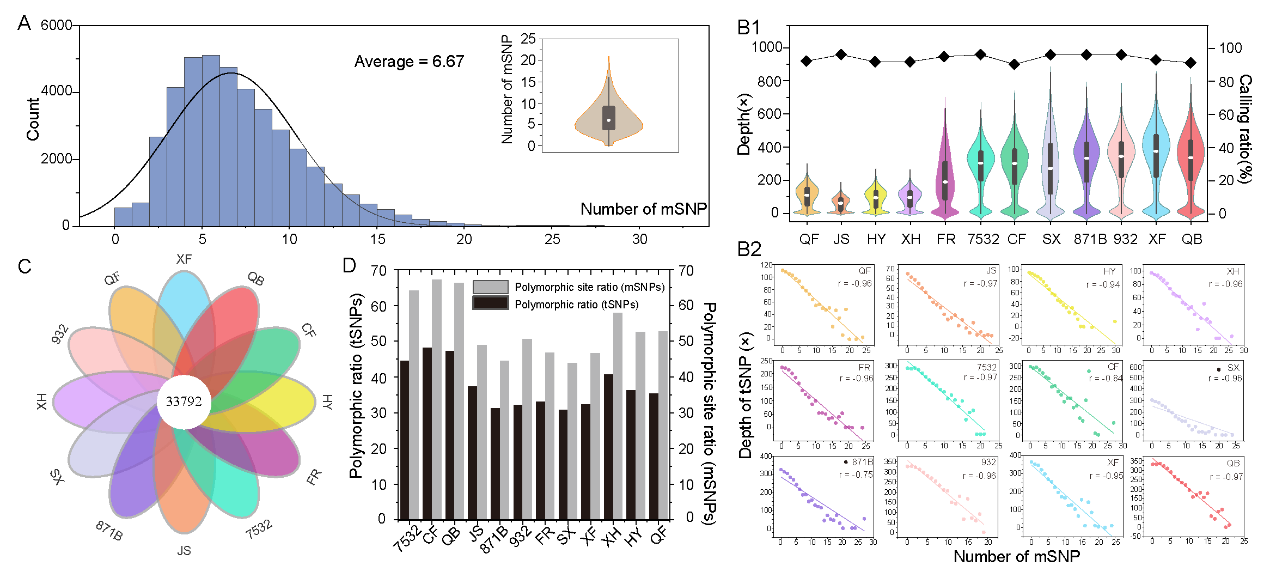
芯片分型能力及多态性水平评估
研究团队利用该芯片对两个回交群体进行基因分型,成功定位到61个与茧丝产量相关的重要数量性状位点(QTL)。尤为引人注目的是,在1号染色体的一个关键QTL区间内,团队鉴定出两个调控茧丝产量的全新功能基因—BmOCIAD1 和BmCOPB2。通过基因编辑技术敲除 BmOCIAD1基因后,雄性的茧层量(CSW)提升了50.00%,雌性提升36.36%;茧层率(CSR)在雄性中提升了13.49%,雌性中提升了19.17% 。为创制超高产家蚕新品种提供了关键的基因靶标。
更重要的是,本研究首次证明了基因组选择在家蚕育种中的重大潜力。基于Silk_40K芯片,团队在回交分离群体和种质资源群体中对全茧量(CSW)和茧层率(CSR)进行了基因组预测,均获得较高的预测准确性,其中种质资源群体中对CSW和CSR的预测准确度分别高达0.866和0.891。这一成果为家蚕茧丝产量、品质等复杂性状的基因组选择改良提供了关键工具支撑,对于通过品种源头创新推动蚕桑产业高质量发展具有重要意义。
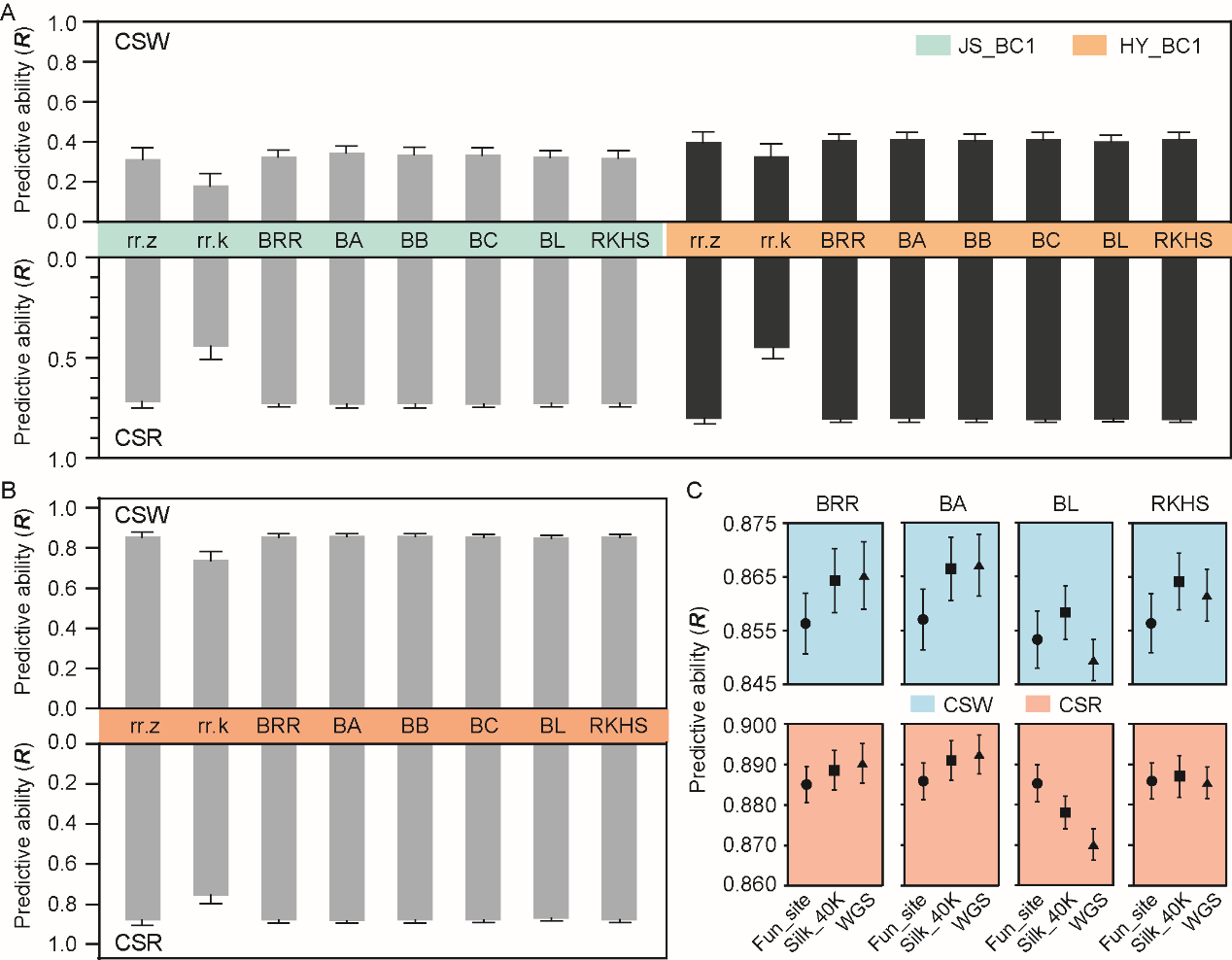
基于Silk_40K Array的家蚕复杂性状基因组预测(茧丝产量为例)
西南大学(资源昆虫高效养殖与利用全国重点实验室、农业农村部蚕桑生物学与遗传育种重点实验室)为该论文第一署名单位,实验室李春林副教授为论文第一作者,代方银教授为通讯作者。该研究得到了国家自然科学基金、国家重点研发计划及重庆市种业创新攻关等项目的支持。
论文链接: https://pubmed.ncbi.nlm.nih.gov/41242493/。
【供稿:李春林; 审核:代方银】